Re: [求救] 請教基因knockdown後影響的相關基因網站
※ 引述《aaaazzzz (找英文家教)》之銘言:
: 各位好,
: 想請教一下有無網站或者相關的microarray data查詢方式,
: 舉例來說,
: 如果想研究EGFR(epidermal growth factor receptor),
: 把這個基因knockdown之後,
: 會造成哪些基因改變?
: 先前曾看過期刊列出microarray data,
: 列出一大堆基因改變的倍數(當然,還需要後續驗證),
: 不曉得有沒有這樣的網站,
: 輸入基因knockdown之後,
: 就會找到對於其他基因的改變(排列由大到小)?
: 或者,各位都是如何去找到期刊microarray的data,
: 來找出基因的相關性?
: 謝謝!
分享我以前搜尋GEO data set的經驗
原po想做的事情跟我以前想做的類似
我當時是蒐集乳癌病人的array找出跟臨床有關係的biomarker
光是蒐集就是個難題
同時要有array資訊也要有臨床資訊
我是採:GEO 資料庫http://www.ncbi.nlm.nih.gov/geo/browse/ 用關鍵字搜尋
原po的關鍵字還不錯 只有3筆,但是運氣也不好:最大的一筆資料只有八個樣本
n=8 ....大概很難作統計吧
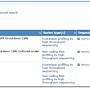
http://imgur.com/g07wG2h

GEO資料庫都有算表達量的結果[關鍵字Series Matrix File(s)]
對生物人是方便的
接下來就是瞭解材料內容,就靠看論文
1.細胞種類 2.組織 3.臨床特性 4.treatment等等
我碩論光蒐集資料就花了一年,請不要小看查論文的過程
我曾經看過有資料庫紀載某種cell line的表達量
但是要有treatment後的表達的資料庫,這種資料庫大概不存在吧
我猜最接近的大概是encode計畫吧?
加油吧,我個人覺得沒有簡單的方式,只能靠自己算
PS 發表這三筆資料的剛好是台灣人洪明奇教授@MD Anderson,滿有意思的
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 122.146.54.185
※ 文章網址: https://www.ptt.cc/bbs/Biotech/M.1458478822.A.B89.html
※ 編輯: lelojack (122.146.54.185), 03/20/2016 21:06:39
→
03/20 21:58, , 1F
03/20 21:58, 1F
→
03/20 22:00, , 2F
03/20 22:00, 2F
→
03/20 22:03, , 3F
03/20 22:03, 3F
→
03/20 22:12, , 4F
03/20 22:12, 4F
→
03/20 22:16, , 5F
03/20 22:16, 5F
→
03/20 22:43, , 6F
03/20 22:43, 6F
→
03/20 22:43, , 7F
03/20 22:43, 7F
→
03/21 00:17, , 8F
03/21 00:17, 8F
推
03/21 23:16, , 9F
03/21 23:16, 9F
→
03/21 23:17, , 10F
03/21 23:17, 10F
推
03/21 23:21, , 11F
03/21 23:21, 11F
→
03/21 23:22, , 12F
03/21 23:22, 12F
→
03/21 23:22, , 13F
03/21 23:22, 13F
→
03/21 23:23, , 14F
03/21 23:23, 14F
→
03/21 23:24, , 15F
03/21 23:24, 15F
→
03/21 23:25, , 16F
03/21 23:25, 16F
→
03/21 23:25, , 17F
03/21 23:25, 17F
→
03/21 23:49, , 18F
03/21 23:49, 18F
→
03/21 23:49, , 19F
03/21 23:49, 19F
→
03/22 01:34, , 20F
03/22 01:34, 20F
→
03/22 01:47, , 21F
03/22 01:47, 21F
→
03/22 01:49, , 22F
03/22 01:49, 22F
→
03/23 00:38, , 23F
03/23 00:38, 23F
推
03/23 22:00, , 24F
03/23 22:00, 24F
→
03/23 22:01, , 25F
03/23 22:01, 25F
→
03/23 22:01, , 26F
03/23 22:01, 26F
推
03/23 22:05, , 27F
03/23 22:05, 27F
→
03/23 22:06, , 28F
03/23 22:06, 28F
→
03/24 11:52, , 29F
03/24 11:52, 29F
推
03/25 17:34, , 30F
03/25 17:34, 30F
→
03/25 20:55, , 31F
03/25 20:55, 31F
→
03/26 00:51, , 32F
03/26 00:51, 32F
討論串 (同標題文章)
本文引述了以下文章的的內容:
完整討論串 (本文為第 2 之 2 篇):